PyTorchでA3C
PyTorchについて
Torchをbackendに持つPyTorchというライブラリがついこの間公開されました.
PyTorchはニューラルネットワークライブラリの中でも動的にネットワークを生成するタイプのライブラリになっていて, 計算が呼ばれる度に計算グラフを保存しておきその情報をもとに誤差逆伝搬します.
動的ネットワークライブラリに対して静的ネットワークライブラリ(tensorflowなど)があります.
静的ネットワークライブラリでは計算グラフの形をまず構築し, そのネットワークに対してデータフィードを行うことで順伝搬, 逆伝搬計算を行います.
動的ネットワークライブラリは入力データに対してネットワーク構造を決めることができるのでRNNや再帰構造をもつネットワークなどが書きやすいという利点があります.
動的ネットワークといえばchainerが有名で, PFNが開発をしています.
今回使用するPyTorchのコードを見てみるとchainerのコードと非常に似通っています.
それもそのはずで
@brandondamos Yep, the PyTorch autograd codebase started with a fork from Chainer -- but then rewrote it in highly optimized pure C
— James Bradbury (@jekbradbury) 2017年1月18日
とあるように自動微分の部分はchainerが元になっているようです.
@chrisemoody @brandondamos @Smerity @TsendeeMTS @ChainerOfficial I’d love to do it eventually (not easy b/c CuPy and Torch are incompatible)
— James Bradbury (@jekbradbury) 2017年1月18日
とありますが, tweetにある通りtorchとcupyの抽象化は大変そうです.
コードを見てみた感じ, forwardとbackwardを定義するのは同じですがlossに対するbackwardの呼び出しがCで書かれていてpythonでbackwardを遡るchainerより速いのかなぁと思ってますが, 深層学習のボトルネックは行列演算とメモリコピーだと思うのでchainerが遡る際に無駄なメモリコピーをしてない限りそこまで速くならない気がしてます(実際そこがchainerの負荷になってるかはわからないので誰か教えてください).
今回PyTrochに注目したのはmultiprocessingがライブラリ内でサポートしてあるので非同期, 分散計算が楽に書けるかも?と思ったからです.
Pythonのmultiprocessing
CPythonではGIL(Global Interpreter Lock)というものを採用していて, 1つのプロセス内で実行できるのは1つのthreadのみとなっています.
そのため複数のコアを使い倒すプログラムを書く時はmultiprocessingモジュールを使用して複数のプロセスを立ち上げる必要があります.
その際複数プロセス間で何かしらの値を共有する必要があります.
multiprocessingモジュールではmultiprocessing.RawArrayを用いると与えた配列を共有メモリ空間上に配置してくれます.
さらにこの配列をnumpyのarrayとして利用したい場合はnumpy.frombufferに確保したRawArrayを渡して共有メモリ空間上に配置されたnumpyのndarrayを得ることができます.
この方法を使ってmultiprocessingなA3C(Asynchronous Advantage Actor-Critic)を実装してある例にmuupanさんのhttps://github.com/muupan/async-rlがあります.
このようにしてmultiprocessingでnumpyを共有することができるんですが, PyTorchではフレームワークで共有をサポートしていて, modelに対してshare_memoryを呼ぶだけでそのmodel内のparameterを共有メモリ上に配置してくれます.
要はRawArray〜, frombuffer〜ってやるのは面倒だし現代的じゃないと思ったのでフレームワークでカバーしてくれるのはいいなと思って試してみました.
A3C
上で紹介したレポジトリにあるA3Cですが, これを再現実装します.https://arxiv.org/abs/1602.01783
強化学習については今回あまり詳しく説明しないのですが, A3Cで重要な部分だけかいつまんで説明します.
A3Cでは各エージェントが各プロセスで行動を行い(報酬, 観測, 行動)の組を集め, その情報を元にローカルのパラメーターに対する勾配を計算し, それを元にグローバルのパラメーターを更新します.
更新後グローバルなパラメーターとローカルなパラメーターは同期され再度rolloutを行います.
共有されるネットワークと各プロセスに存在するローカルなネットワークで通信が行われます.
実装
まずニューラルネットワークを実装します.
A3Cでは方策関数と状態価値観数をニューラルネットワークで表現します.
PyTorchではnn.Moduleを継承したクラスのforwardを定義してあげるとcallableなLayerを作ることができます.
class Policy(nn.Module): def __init__(self, num_actions, dim_obs, frame_num=4): super(Policy, self).__init__() self.num_actions = num_actions self.dim_obs = dim_obs self.frame_num = frame_num self.fc1 = nn.Linear(dim_obs*frame_num, 128) self.fc2 = nn.Linear(128, 128) self.p = nn.Linear(128, num_actions) self.v = nn.Linear(128, 1) def forward(self, x): x = F.relu(self.fc1(x)) x = F.relu(self.fc2(x)) policy = self.p(x) value = self.v(x) return F.softmax(policy), value def sync(self, global_module): for p, gp in zip(self.parameters(), global_module.parameters()): p.data = gp.data.clone()
このpolicyをglobalとlocalで2つ宣言してあげます.
global_policyは共有メモリ上に配置します.
global_policy = Policy(env.action_space.n, env.observation_space.shape[0], args.frame_num) global_policy.share_memory() local_policy = Policy(env.action_space.n, env.observation_space.shape[0], args.frame_num)
このあと各プロセスを起動して非同期更新を行います.
勾配計算はtrain関数内で行われます.
def train(rank, global_policy, local_policy, optimizer, env, global_t, args): o = env.reset() step = 0 sum_rewards = 0 max_sum_rewards = 0 while global_t[0] < args.epoch: local_policy.sync(global_policy) observations = [] actions = [] values = [] rewards = [] probs = [] R = 0 for i in range(args.local_t_max): global_t += 1 step += 1 p, v = local_policy(o) a = p.multinomial() o, r, done, _ = env.step(a.data.squeeze()[0]) if rank == 0: sum_rewards += r if args.render: env.render() observations.append(o) actions.append(a) values.append(v) rewards.append(r) probs.append(p) if done: o = env.reset() if rank == 0: print('----------------------------------') print('total reward of the episode:', sum_rewards) print('----------------------------------') if args.save_mode == 'all': torch.save(local_policy, os.path.join(args.log_dir, args.save_name+"_{}.pkl".format(global_t[0]))) elif args.save_mode == 'last': torch.save(local_policy, os.path.join(args.log_dir, args.save_name+'.pkl')) elif args.save_mode == 'max': if max_sum_rewards < sum_rewards: torch.save(local_policy, os.path.join(args.log_dir, args.save_name+'.pkl')) max_sum_rewards = sum_rewards sum_rewards = 0 step = 0 break else: _, v = local_policy(o) R += v.data.squeeze()[0] returns = [] for r in rewards[::-1]: R = r + 0.99 * R returns.insert(0, R) returns = torch.Tensor(returns) if len(returns) > 1: returns = (returns-returns.mean()) / (returns.std()+args.eps) v_loss = 0 entropy = 0 for a, v, p, r in zip(actions, values, probs, returns): a.reinforce(r - v.data.squeeze()) _v_loss = nn.MSELoss()(v, Variable(torch.Tensor([r]))) v_loss += _v_loss entropy += (p * (p + args.eps).log()).sum() v_loss = v_loss * 0.5 * args.v_loss_coeff entropy = entropy * args.entropy_beta optimizer.zero_grad() final_node = [v_loss, entropy] + actions gradients = [torch.ones(1), torch.ones(1)] + [None] * len(actions) autograd.backward(final_node, gradients) new_lr = (args.epoch - global_t[0]) / args.epoch * args.lr optimizer.step(new_lr)
余談なんですが, このコードやPyTorchのexampleshttps://github.com/pytorch/examples/でのreinforce.pyやactor-critic.pyで使われているmethodにreinforceがあります.
reinforceはREINFORCEができます.
REINFORCEは確率分布に対して誤差逆伝搬法を使いたい時に使う手法で, サンプリングによって微分ができなくなる際に勾配を推定する手法です.
PyTorchではStochasticFunctionというクラスがあり, そこからサンプリングされたものに対してはREINFORCEを使うことができます.
これはlog(p)をlossにすればいいだけの話なんですが, こっちのほうがわかりやすくていいと思います.
最後はこのtrainをmultiprocessingで並列実行すると非同期に学習してくれます.
processes = []
for rank in range(args.num_process):
p = mp.Process(target=train, args=(rank, global_policy, local_policy, optimizer, env, global_t, args))
p.start()
processes.append(p)
for p in processes:
p.join()
結果
とりあえずCartPoleだけ試してみました.

まぁまぁ学習してそうでしょうか?
今回のコードとか
コードは全部https://github.com/rarilurelo/pytorch_a3cにあります.
PyTorchのインストールは本家ページhttp://pytorch.org/の通りにやるとすぐできます.
あとがき
今日ちょうど秋葉さんがchainerを分散並列で高速化した話https://www.youtube.com/watch?v=wPr-yuJjvFQを見てました.
その中でコミュニティの認識としては非同期更新より同期更新がやはりいいと言うことが述べられていました.
実際A3Cをfxデータに適用したことがあるんですが, 途中まではうまい感じで学習しても方策が壊れてしまうことが多々ありハイパラ調整は難しいです.
A3CがOn-Policyの学習アルゴリズムであることも関係していると思われるのですが, 一回変な方策ができるとそこから回復することはありません.
TRPOhttps://arxiv.org/abs/1502.05477では方策を崩さずに単調な増加を目指すために色々頑張っているよう思うのですがTRPOの強さを考えると強化学習の難しい課題なのではないかと思います.
脳のように非同期学習を行うニューラルネットワークの実装 with keras tensorflow backend
はじめに
タイトルには脳の非同期学習というようにまるで脳が非同期的に学習をしているかのように書きましたが, そこんところ実際はどうなっているかよくわかりません.
自転車を漕ぎながら考えごとをしたり, サッカーでドリブルしながらシュートかパスか考えたり, 少なくとも思考と運動の処理自体は並列非同期であるよう私自身は思います.
まぁよくわからないんですが, とりあえずニューラルネットワークでは非同期でも学習できたよ! というのが今回紹介する論文です.
どんなことをしているか
まず言葉の意味から行くと, Decoupledとは分離という意味です.
なにを分離するのかというと層の依存関係を分離します.
ニューラルネットワークでは, 当たり前なのですが, 次の層に渡る前に前の層で値が計算される必要があります.
これをForward Lockingといいます.
順伝搬方向に並列化つまり分離して非同期に処理を行うことができないということです.
また, 層の更新も誤差の伝播を待つ必要があり, Update Lockingといいます.
誤差自体の計算も計算するには上の層での計算を待たなければならないのでこれをBackward Lockingといいます.
これら3つの依存関係のうち, Backward Lockingを分離したのが論文内でDNI(Decoupled Neural Interfaces)と呼ばれる層の結合関係です(実はForward Lockingを分離したモデルも提案している)
DNI
論文中にかなりざっくりとした近似式で書かれていますが, 勾配の計算を上の層全て使うのではなく現在の層が出した値で近似してしまえば上からの誤差なんて待たずにすむやんって感じです.
そして, 近似といえばニューラルネットワークです.
ニューラルネットワークを学習するための勾配を生成するニューラルネットワーク(論文内でM)を構築してやることでこの近似を成り立たせBackward Lockingを分離します.
ちなみにM自体の学習には1つ上のMが出した誤差を使って学習するので, 1個上の層の計算を待つだけで更新ができます
この学習の過程はDeepMindのアニメーションがすごいわかりやすいです.
実験
実験は通常のFeedForwardネットワークに加え, rnn対して行っています.
特にrnnは通常であればtruncateする部分でMが出した誤差を使うことで収束性能の向上や, DNC(Differentiable Neural Computers)の前身であるNTM(Neural Turing Machine)で行われていた記号列の記憶タスクで良い結果を出しています.
特におもしろいのがMultiNetworkSystemに関する実験で, 一風変わったタスクを解いています.
流れてくる数字をrnnで構成されたネットワークAで受取り, T回に一回, 受け取った数字の内奇数の個数を出力する, かつ, ネットワークBに隠れ層を渡します.
渡されたBはT^2回に一回流れてきた数字の内「3」の個数を出力します.
従来の誤差逆伝搬法ではT^2回に一回しか誤差が計算されず, かといってAのタスクだけで更新するとBに必要な情報が取れなくなります.
そこでBに順伝搬する時にMを使うことで収束を速め, かつ精度を維持しています.
単純な入出力だけでなく異なるタスク間で通信を行うネットワーク構造は, 入力に対して考えを巡らせる人間のようでおもしろいです.
詳しい実験結果
以前輪読会で発表したときにまとめた資料があるのでそれを観ると上の説明ではよくわからなかった部分もわかるかもしれません, よりわからなくなるかもしれません.
追実験
正直DeepMindから出ているとはいえ, 実際の勾配を使わずに学習できるのか信じがたいので再実験をすることにしました.
一方向のネットワークを実装し, MNISTの分類タスクに適用しました.
https://github.com/rarilurelo/tensorflow-synthetic_gradient
このページにあるようにちゃんと学習できています(少し収束が遅いですが).
実装
勾配に手を加える実装は通常のフレームワークを使用していると難しいところです.
ほとんどのフレームワークは勾配計算を自動で行ってくれてしまうからです.
幸いtensorflowにはtf.gradientsという関数が準備されていて勾配の操作を行うことができます.
この関数は引数にobjective functionと勾配を計算したいパラメタを入力することでそのパラメタに対する勾配を自動で計算してくれます.
これに加え, 勾配の初期値をgrad_ys引数を使って設定することができます.
まずLayerを順伝搬の分とM(cDNI)の分で定義します.
# Layer1 layer1 = Sequential() layer1.add(Dense(256, input_dim=784)) layer1.add(BatchNormalization(mode=2)) layer1.add(Activation('relu')) # Layer2 layer2 = Sequential() layer2.add(Dense(256, input_dim=256)) layer2.add(BatchNormalization(mode=2)) layer2.add(Activation('relu')) # Layer3 layer3 = Sequential() layer3.add(Dense(256, input_dim=256)) layer3.add(BatchNormalization(mode=2)) layer3.add(Activation('relu')) # Layer4 layer4 = Sequential() layer4.add(Dense(10, activation='softmax', input_dim=256)) # cDNI1 belongs to Layer2, so it accepts Layer1's output and emmits grad_y of Layer1 cDNI1 = Sequential() cDNI1.add(Dense(1024, input_dim=256+10)) cDNI1.add(BatchNormalization(mode=2)) cDNI1.add(Activation('relu')) cDNI1.add(Dense(1024)) cDNI1.add(BatchNormalization(mode=2)) cDNI1.add(Activation('relu')) cDNI1.add(Dense(256, weights=[np.zeros(shape=[1024, 256])], bias=False)) # cDNI2 belongs Layer3 cDNI2 = Sequential() cDNI2.add(Dense(1024, input_dim=256+10)) cDNI2.add(BatchNormalization(mode=2)) cDNI2.add(Activation('relu')) cDNI2.add(Dense(1024)) cDNI2.add(BatchNormalization(mode=2)) cDNI2.add(Activation('relu')) cDNI2.add(Dense(256, weights=[np.zeros(shape=[1024, 256])], bias=False)) # cDNI3 belongs Layer4 cDNI3 = Sequential() cDNI3.add(Dense(1024, input_dim=256+10)) cDNI3.add(BatchNormalization(mode=2)) cDNI3.add(Activation('relu')) cDNI3.add(Dense(1024)) cDNI3.add(BatchNormalization(mode=2)) cDNI3.add(Activation('relu')) cDNI3.add(Dense(256, weights=[np.zeros(shape=[1024, 256])], bias=False))
Layer1の出した値をLayer2に伝搬して同時にMで勾配を算出します.
# layer1の算出した値y_l1をlayer2の入力x_l2に代入 x_l2 = y_l1 # layer2の順伝搬 y_l2 = layer2(x_l2) # y_l2とlabelをlayer2のMに入れて勾配を算出 p_gy_l2 = cDNI2(K.concatenate((y_l2, labels), axis=1)) # layer1のMのためにlayer2のMが出した勾配をlayer1に逆伝搬 gy_l1 = tf.gradients(y_l2, y_l1, grad_ys=p_gy_l2)[0] # layer1のMのlossfunction loss_dni1 = K.mean(K.sum((p_gy_l1-gy_l1)**2, 1)) # layer1のMのパラメタへの勾配を計算 grad_trainable_weights_dni1 = tf.gradients(loss_dni1, cDNI1.trainable_weights) # layer2のMが算出した勾配を使ってlayer2のパラメタへの勾配を計算 grad_trainable_weights_l2 = tf.gradients(y_l2, layer2.trainable_weights, grad_ys=p_gy_l2)
この操作を各層に渡って行って勾配を計算して更新します.
この時注意するのが全層での勾配の計算が終わってから層を更新する必要があるということです.
tensorflowでは計算グラフに対してtensorをfeedしていくのですが, その計算順序は固定されずノードに値が来たら計算するだけなのでlayerの更新が行われてから勾配計算が行われたりすると不都合です.
そのためtf.control_dependenciesを使って計算依存性をつけなければなりません.
with tf.control_dependencies(gparams):
updates = optimizer.get_updates(params, gparams)
またtensorflowでは非同期連続でデータを与えることができません(QUEUEがどうなっているのかは知りません).
なので結局非同期更新はできないのですが, このコードで結果をシミュレーションできます.
実装の全体はhttps://github.com/rarilurelo/tensorflow-synthetic_gradientに公開しています.
まとめ
Neural Networkの非同期学習を実装しました.
こうして実際に動いているところをみると勾配をわざわざ微分して求めることはすこし大袈裟なのではないかとすら思えます.
神経科学的妥当性も気になるところです.
非同期にすることで学習を並列に動かし学習を劇的に速くできる可能性があります.
しかし現状のフレームワークでdata feedを並列非同期で行えるのはchainerを改造するぐらいしか思いつきません.(chainerはそもそもdata feedという概念がない)
マルチスレッドでlayerにGPUを割り当てたらできそうな気がするので時間があったら実装してみたいです.
今後Neural Networkの学習がどんな発展をするかまったく予測はできませんが, 前提とされている計算順序なども壊されてくるのならより柔軟な操作が必要になるのでしょう.
VAEからCVAE with keras
はじめに
出てきた当初は画像分類タスクで猛威を振るった深層学習ですが, 最近はいろんな機械学習と組み合わせで応用されています. 強化学習を応用したAlphaGoでイ・セドルを打ち負かしたり, 画像認識と自然言語処理の組み合わせで画像のキャプションを生成したり, 生成モデルに応用して自然に近い画像を作るなど賑わいを見せています.
今回は画像生成手法のうちのDeepLearningを自然に生成モデルに拡張したと考えられるVAE(Variational Auto Encoder)から, その発展系であるCVAE(Conditional VAE)までを以下2つの論文をもとに自分の書いたkerasのコードとともに紹介したいと思います.
Auto-Encoding Variational Bayes
Semi-Supervised Learning with Deep Generative Models
目次
VAE系手法の良い所
確率分布をニューラルネットワークを用いて表現できると次の2つの良い点があります.
- サンプリングによって新たな画像(学習画像の生成分布に沿った)を生成することができる
- 潜在空間の変数をいじることによって狙った画像を生成したり画像どうしのアナロジーをみることができる
この2つの点はそのまま生成モデルの良い点だということができます. そこでまずは生成モデルとはなんなのか自分なりにまとめてみます.
生成モデル
抽象的な話をすると生成モデルでは観測されたデータに対して, 非観測な潜在変数
を仮定し,
が確率分布
からサンプリングされ, 生成された
を用いて
から
が生成されたとして数理モデルに落とし込みます.
つまりそこにあるデータの背後にはなにか見えない変数が存在しそれをもとにデータが生成されたとする一種の考え方とも言えます.
具体的な例として高3男子の身長の分布を考えてみます. その分布の形状は正規分布に近い形になっていることが知られているので今回は正規分布を仮定して考えます. 確率分布は形状とパラメータで定まります. 形状は正規分布を仮定しているのでパラメータと
を使って
となります.
このと
をただのパラメータではなく確率変数(何かの分布に従っている)として
と
を潜在変数とした生成モデルを考えることができます.
と
には上の
にあたる事前分布が存在します.
図で書くと下のようになります.

生成モデルではサンプリングを行えるので高3男子の身長のサンプルがほしい!となったらと
の事前分布から
と
を生成し
からサンプリングすればよいわけです. (このようなシチュエーションがあるかはしらない)
さらには確率を計算できるので適当な身長をの
に代入してその身長が高3男子としてどれだけ確からしいかを数値として割り出すこともできます.
VAEとは
VAEでは生成モデルの枠組みをそのまま利用します. 学習するデータ(画像など)に対して潜在変数を仮定し
から
を生成し,
で
が生成されたとします.
このをNeuralNetworkで表現します.
確率分布をNNで表す
VAEでは確率分布のgivenな変数(でいうところの
)を引数に取り, 確率分布のパラメータを出力にするNeuralNetworkで確率分布を表現します. 形状はprioriで実験ではガウス分布やベルヌーイ分布を使用しています.
具体的にガウス分布でmnist(784次元)を考えてみるとを入力にとって784次元の
ベクトルと
ベクトルを出力します. 各次元に対して一次元ガウス分布のサンプリングを行うことで
を生成できます.
確率分布の実装
確率分布のNNによる表現方法はきまったのでそれをどのように実装するかという話になります.
今回の実装ではkerasを使用したのでkerasのfunctionalAPIの機能をふんだんに使用しました.
functionalAPIについて詳しくはkeras tutorialを見てください.
Sequentialを使うことで入力, 出力をtensorに持つNeuralNetworkを1つの関数のように構築できます.
NeuralNetworkで表される条件付き確率分布の主要な機能としては,
- givens(
)が与えられた時
- サンプリング
- パラメタ計算
- givens(
)と確率変数(
)が与えられた時
- 確率の計算
- 対数尤度の計算
ができればよいのでそれをみたすクラスを作りました. initの引数でkerasのSequentialオブジェクトを与えます.
class GaussianDistribution(ProbabilityDistribution): def __init__(self, variable, givens=None, mean=0, var=1, mean_model=None, var_model=None): self.variable = variable self.variable_shape = K.int_shape(self.variable) def sample(args): mean, var = args epsilon = K.random_normal(K.shape(mean)) return mean+var*epsilon self.draw = Lambda(sample) self.mean_model = mean_model self.var_model = var_model if givens is None: if isinstance(mean, float) or isinstance(mean, int): self.mean = K.ones_like(self.variable)*mean else: self.mean = mean if isinstance(var, float) or isinstance(var, int): self.var = K.ones_like(self.variable)*var else: self.var = var def get_params(self, givens=None): if givens is None: return self.mean, self.var mean = self.mean_model(givens) var = self.var_model(givens) return mean, var def sampling(self, givens=None): if givens is None: return self.draw([self.mean, self.var]) mean = self.mean_model(givens) var = self.var_model(givens) return self.draw([mean, var]) def prob(self, variable, givens=None): if givens is None: return 1/K.sqrt(2*np.pi*self.var)*K.exp(-1/2*(variable-self.mean)**2/self.var) mean = self.mean_model(givens) var = self.var_model(givens) return 1/K.sqrt(2*np.pi*var)*K.exp(-1/2*(variable-mean)**2/var) def _log_gausian(self, variable, mean, var): return -1/2*K.log(K.clip(2*np.pi*var, K._epsilon, 1/K._epsilon))-1/2*(variable-mean)**2/K.clip(var, K._epsilon, 1/K._epsilon) def logliklihood(self, variable, givens=None): """ a mean logliklihood of minibatch """ if givens is None: return K.mean(K.sum(self._log_gausian(variable, self.mean, self.var), axis=1)) mean = self.mean_model(givens) var = self.var_model(givens) return K.mean(K.sum(self._log_gausian(variable, mean, var), axis=1))
このクラスを使って実装していきます.
cost関数
実装を始める前にcost関数を定める必要があります.
生成モデルでのコスト関数がであることは問題ないと思います. 作った確率分布に対して, 実際のトレーニングデータ
の生成される確率を計算し, その値が大きいほどその生成分布は実際のデータに即していると考えられるからです. ここでは負の対数を取ってあります.
このを式変形していき, 計算できる値まで持って行きます.
(1)は周辺化の定義式で, (2)はベイズの定理, (3)はの導入, (4)はイェンゼンの不等式, (6)は期待値とKLダイバージェンスの定義式を利用して式変形しています.
急に出てきたは代理分布と呼ばれ,
の計算が困難な場合に用いられます.
第一項の期待値はからサンプリングした
個の点を用いて
で近似できます. 論文中では大きなミニバッチサイズで計算することで
でも良い結果が得られることが書いてあります.
第二項のKLダイバージェンスは解析的に計算することができ, となります.
この辺の式変形はAuto-Encoding Variational BayesのAppendixに詳しく書いてあるのと, 日本語の記事ではVariational Dropout and the Local Reparameterization Trickや正規分布間のKLダイバージェンスの導出を参考にしました.
実装
先ほど作ったNNを持つ確率分布のclassを使ってVAEclassを実装します.
確率分布がもつNNの構造をkerasのSequentialで記述し確率分布を生成し, その確率分布をもとにのサンプリングと
の復元を行いcostを計算します.
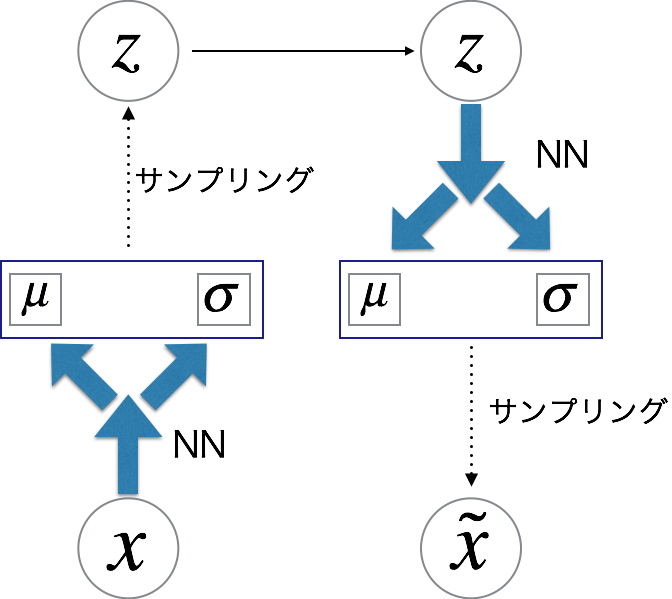
class VAEM1(object): def __init__(self, in_dim=784, hid_dim=300, z_dim=50): self.in_dim = in_dim self.hid_dim = hid_dim self.z_dim = z_dim self.x = Input((self.in_dim, )) self.z = Input((self.z_dim, )) ############ # q(z | x) # ############ model = Sequential() model.add(Dense(self.hid_dim, input_dim=self.in_dim)) model.add(CustomBatchNormalization()) model.add(Activation('softplus')) model.add(Dense(self.hid_dim)) model.add(CustomBatchNormalization()) model.add(Activation('softplus')) mean = Sequential([model]) mean.add(Dense(self.hid_dim)) mean.add(CustomBatchNormalization()) mean.add(Activation('softplus')) mean.add(Dense(self.z_dim)) var = Sequential([model]) var.add(Dense(self.hid_dim)) var.add(CustomBatchNormalization()) var.add(Activation('softplus')) var.add(Dense(self.z_dim, activation='softplus')) self.q_z_x = GaussianDistribution(self.z, givens=[self.x], mean_model=mean, var_model=var) ############ # p(x | z) # ############ model = Sequential() model.add(Dense(self.hid_dim, input_dim=self.z_dim)) model.add(CustomBatchNormalization()) model.add(Activation('softplus')) model.add(Dense(self.hid_dim)) model.add(CustomBatchNormalization()) model.add(Activation('softplus')) model.add(Dense(self.in_dim, activation='sigmoid')) self.p_x_z = BernoulliDistribution(self.x, givens=[self.z], model=model) ######################## #sample and reconstruct# ######################## self.sampling_z = self.q_z_x.sampling(givens=[self.x]) self.reconstruct_x = self.p_x_z.sampling(givens=[self.sampling_z]) def _KL(self, mean, var): return -1/2*K.mean(K.sum(1+K.log(K.clip(var, K._epsilon, 1/K._epsilon))-mean**2-var, axis=1)) def cost(self, inputs, outputs): mean, var = self.q_z_x.get_params(givens=[self.x]) KL = self._KL(mean, var) logliklihood = self.p_x_z.logliklihood(self.x, givens=[self.sampling_z]) lower_bound = -KL+logliklihood lossfunc = -lower_bound return lossfunc def training_model(self): model = Model(input=self.x, output=self.reconstruct_x) return model def encoder(self): model = Model(input=self.x, output=self.sampling_z) return model def decoder(self): decode = self.p_x_z.sampling(givens=[self.z]) model = Model(input=self.z, output=decode) return model
トレーニングコードは以下のようになります.
from __future__ import division from keras.datasets import mnist from vae_m1 import VAEM1 nb_epoch = 30 if __name__ == '__main__': (X_train, y_train), (X_test, y_test) = mnist.load_data() X_train = X_train.reshape(-1, 28*28) X_test = X_test.reshape(-1, 28*28) X_train = X_train/255.0 X_test = X_test/255.0 X_train[X_train > 0.5] = 1.0 X_train[X_train <= 0.5] = 0.0 X_test[X_test > 0.5] = 1.0 X_test[X_test <= 0.5] = 0.0 vaem1 = VAEM1() training = vaem1.training_model() training.compile(optimizer='adam', loss=vaem1.cost) training.fit(X_train, X_train, batch_size=100, nb_epoch=nb_epoch, shuffle=True, validation_data=(X_test, X_test) ) encoder = vaem1.encoder() encoder.save('./trained_model/encoder_m1.h5') decoder = vaem1.decoder() decoder.save('./trained_model/decoder_m1.h5')
CVAEとは
CVAEでは隠れ変数の1つにlabelを加えた確率モデルを考えます.
Semi-Supervised Learning with Deep Generative Models中でM2モデルと書かれている物です. 生成モデルとしてはを学習したいのですがcost関数が少し変化します.
これがlabel付きデータのloss関数になります. 論文中で半教師あり学習を行う際にラベルなしデータに対するloss関数を導出していますが, 今回半教師あり学習は行わずにアナロジーだけ見るので解説はしません. 実装はしてあるので見てみてください.
論文中ではこのM2モデルにVAEで出てきた潜在変数を入力変数として適用することでstacked VAEとしています.
VAEM2モデルのclass実装は下のようになります.
class VAEM2(object): def __init__(self, in_dim=50, cat_dim=10, hid_dim=300, z_dim=50, alpha=0): self.in_dim = in_dim self.cat_dim = cat_dim self.hid_dim = hid_dim self.z_dim = z_dim self.alpha = alpha self.x_l = Input((self.in_dim, )) self.x_u = Input((self.in_dim, )) self.y_l = Input((self.cat_dim, )) y_u0 = Input((self.cat_dim, )) y_u1 = Input((self.cat_dim, )) y_u2 = Input((self.cat_dim, )) y_u3 = Input((self.cat_dim, )) y_u4 = Input((self.cat_dim, )) y_u5 = Input((self.cat_dim, )) y_u6 = Input((self.cat_dim, )) y_u7 = Input((self.cat_dim, )) y_u8 = Input((self.cat_dim, )) y_u9 = Input((self.cat_dim, )) self.y_u = [y_u0, y_u1, y_u2, y_u3, y_u4, y_u5, y_u6, y_u7, y_u8, y_u9] self.z = Input((self.z_dim, )) ############### # q(z | x, y) # ############### x_branch = Sequential() x_branch.add(Dense(self.hid_dim, input_dim=self.in_dim)) x_branch.add(CustomBatchNormalization()) x_branch.add(Activation('softplus')) y_branch = Sequential() y_branch.add(Dense(self.hid_dim, input_dim=self.cat_dim)) y_branch.add(CustomBatchNormalization()) y_branch.add(Activation('softplus')) merged = Sequential([Merge([x_branch, y_branch], mode='concat')]) merged.add(Dense(self.hid_dim)) merged.add(CustomBatchNormalization()) merged.add(Activation('softplus')) mean = Sequential([merged]) mean.add(Dense(self.hid_dim)) mean.add(CustomBatchNormalization()) mean.add(Activation('softplus')) mean.add(Dense(self.z_dim)) var = Sequential([merged]) var.add(Dense(self.hid_dim)) var.add(CustomBatchNormalization()) var.add(Activation('softplus')) var.add(Dense(self.z_dim, activation='softplus')) self.q_z_xy = GaussianDistribution(self.z, givens=[self.x_l, self.y_l], mean_model=mean, var_model=var) ############### # p(x | y, z) # ############### y_branch = Sequential() y_branch.add(Dense(self.hid_dim, input_dim=self.cat_dim)) y_branch.add(CustomBatchNormalization()) y_branch.add(Activation('softplus')) z_branch = Sequential() z_branch.add(Dense(self.hid_dim, input_dim=self.z_dim)) z_branch.add(CustomBatchNormalization()) z_branch.add(Activation('softplus')) merged = Sequential([Merge([y_branch, z_branch], mode='concat')]) merged.add(Dense(self.hid_dim)) merged.add(CustomBatchNormalization()) merged.add(Activation('softplus')) mean = Sequential([merged]) mean.add(Dense(self.hid_dim)) mean.add(CustomBatchNormalization()) mean.add(Activation('softplus')) mean.add(Dense(self.in_dim)) var = Sequential([merged]) var.add(Dense(self.hid_dim)) var.add(CustomBatchNormalization()) var.add(Activation('softplus')) var.add(Dense(self.in_dim, activation='softplus')) self.p_x_yz = GaussianDistribution(self.x_l, givens=[self.y_l, self.z], mean_model=mean, var_model=var) ######## # p(y) # ######## self.p_y = CategoricalDistribution(self.y_l) ############ # q(y | x) # ############ inference = Sequential() inference.add(Dense(self.hid_dim, input_dim=self.in_dim)) inference.add(CustomBatchNormalization()) inference.add(Activation('softplus')) inference.add(Dense(self.hid_dim)) inference.add(CustomBatchNormalization()) inference.add(Activation('softplus')) inference.add(Dense(self.cat_dim, activation='softmax')) self.q_y_x = CategoricalDistribution(self.y_l, givens=[self.x_l], model=inference) ########################## # sample and reconstruct # ########################## self.sampling_z = self.q_z_xy.sampling(givens=[self.x_l, self.y_l]) self.reconstruct_x_l = self.p_x_yz.sampling(givens=[self.y_l, self.sampling_z]) def _KL(self, mean, var): return -1/2*K.mean(K.sum(1+K.log(K.clip(var, K._epsilon, 1/K._epsilon))-mean**2-var, axis=1)) def label_cost(self, y_true, y_false): ########### # Labeled # ########### self.mean, self.var = self.q_z_xy.get_params(givens=[self.x_l, self.y_l]) KL = self._KL(self.mean, self.var) logliklihood = -self.p_x_yz.logliklihood(self.x_l, givens=[self.y_l, self.sampling_z])-self.p_y.logliklihood(self.y_l) L = KL+logliklihood L = L+self.alpha*self.q_y_x.logliklihood(self.y_l, givens=[self.x_l]) return L def cost(self, y_true, y_false): ########### # Labeled # ########### self.mean, self.var = self.q_z_xy.get_params(givens=[self.x_l, self.y_l]) KL = self._KL(self.mean, self.var) logliklihood = -self.p_x_yz.logliklihood(self.x_l, givens=[self.y_l, self.sampling_z])-self.p_y.logliklihood(self.y_l) L = KL+logliklihood L = L+self.alpha*self.q_y_x.logliklihood(self.y_l, givens=[self.x_l]) ############# # UnLabeled # ############# U = 0 # marginalization for y in self.y_u: mean, var = self.q_z_xy.get_params(givens=[self.x_u, y]) sampling_z = self.q_z_xy.sampling(givens=[self.x_u, y]) U += self.q_y_x.prob(y, givens=[self.x_u])*(-self.p_x_yz.logliklihood(self.x_u, givens=[y, sampling_z]) -self.p_y.logliklihood(y) +self._KL(mean, var) +self.q_y_x.logliklihood(y, givens=[self.x_u]) ) return U+L def label_training_model(self): model = Model(input=[self.x_l, self.y_l], output=self.reconstruct_x_l) return model def training_model(self): model = Model(input=[self.x_l, self.y_l, self.x_u]+self.y_u, output=self.reconstruct_x_l) return model def encoder(self): model = Model(input=[self.x_l, self.y_l], output=self.mean) return model def decoder(self): decode = self.p_x_yz.sampling(givens=[self.y_l, self.z]) model = Model(input=[self.y_l, self.z], output=decode) return model def classifier(self): inference = self.q_y_x.get_params(givens=[self.x_l]) model = Model(input=self.x_l, output=inference) return model
実験
VAE
VAEモデルにmnistを入力して再構成を行いました.
実行コードは下のようになります.
kerasのdefaultのBatchNormalizationはtestの時もbatch単位計算してしまうmodeか, 層を再利用できないmodeしかなかったのでCustomBatchNormalizationという名前で再利用可能かつtest時に今までの平均と分散の指数移動平均を使用する層を作成しました.
from keras.models import load_model from keras.datasets import mnist import matplotlib.pyplot as plt from custom_batchnormalization import CustomBatchNormalization custom_objects={'CustomBatchNormalization': CustomBatchNormalization} if __name__ == '__main__': (X_train, y_train), (X_test, y_test) = mnist.load_data() X_train = X_train.reshape(-1, 28*28) X_test = X_test.reshape(-1, 28*28) X_train = X_train/255.0 X_test = X_test/255.0 X_train[X_train > 0.5] = 1.0 X_train[X_train <= 0.5] = 0.0 X_test[X_test > 0.5] = 1.0 X_test[X_test <= 0.5] = 0.0 encoder = load_model('./trained_model/encoder_m1.h5', custom_objects=custom_objects) decoder = load_model('./trained_model/decoder_m1.h5', custom_objects=custom_objects) targets = X_train[0:5] latents = encoder.predict(targets, batch_size=5) reconstruct_images = decoder.predict(latents, batch_size=5) fig = plt.figure(figsize=(14, 14)) for i, target in enumerate(targets): ax = fig.add_subplot(2, 5, i+1, xticks=[], yticks=[]) ax.imshow(target.reshape(28, 28), 'gray') for i, reconstruct_image in enumerate(reconstruct_images): ax = fig.add_subplot(2, 5, 6+i, xticks=[], yticks=[]) ax.imshow(reconstruct_image.reshape(28, 28), 'gray') plt.savefig('./images/reconstruct_m1.png')
上が元画像で下が再構成画像です. ある程度再構成できています.

数字間アナロジー
上での多様体学習の様子を見てみます. target画像を2つ用意し, その2つを潜在変数空間上に落とし込み潜在空間上に直線を引いて直線上の
を再構成します.
from keras.models import load_model from keras.datasets import mnist import numpy as np import matplotlib.pyplot as plt from custom_batchnormalization import CustomBatchNormalization custom_objects = {'CustomBatchNormalization': CustomBatchNormalization} if __name__ == '__main__': (X_train, y_train), (X_test, y_test) = mnist.load_data() X_train = X_train.reshape(-1, 28*28) X_test = X_test.reshape(-1, 28*28) X_train = X_train/255.0 X_test = X_test/255.0 X_train[X_train > 0.5] = 1.0 X_train[X_train <= 0.5] = 0.0 X_test[X_test > 0.5] = 1.0 X_test[X_test <= 0.5] = 0.0 encoder = load_model('./trained_model/encoder_m1.h5', custom_objects=custom_objects) decoder = load_model('./trained_model/decoder_m1.h5', custom_objects=custom_objects) target1 = X_train[0:1] target2 = X_train[8:9] latent1 = encoder.predict(target1, batch_size=1) latent2 = encoder.predict(target2, batch_size=1) fig = plt.figure(figsize=(14, 14)) for i, d in enumerate(np.linspace(0, 1, 10)): latent = latent1+d*(latent2-latent1) reconstruct_image = decoder.predict(latent, batch_size=1) ax = fig.add_subplot(1, 10, i+1, xticks=[], yticks=[]) ax.imshow(reconstruct_image.reshape(28, 28), 'gray') plt.savefig('./images/analogy_m1.png')

5から1への変化が確認できます.
CVAE
論文中にあるM1モデル(VAE)との併用で実験を行います. VAEを使って全てのmnistトレーニングデータをの潜在空間に持って行きます. 次に潜在空間上のトレーニングデータを入力としてM2モデルをトレーニングします.

VAEの時と同様に再構成してみます.

再構成できています.
筆跡アナロジー
論文中では潜在変数にを追加したことによって
が筆跡のようなものを捉えるようになったと書かれています.
実際に確認するために画像をの潜在変数空間に持って行き固定したまま, ラベル
を0~9で変化させて再構成します.
コードは以下になります.
from keras.models import load_model from keras.datasets import mnist from keras.utils import np_utils import numpy as np import matplotlib.pyplot as plt from custom_batchnormalization import CustomBatchNormalization custom_objects = {'CustomBatchNormalization': CustomBatchNormalization} if __name__ == '__main__': (X_train, y_train), (X_test, y_test) = mnist.load_data() X_train = X_train.reshape(-1, 28*28) X_test = X_test.reshape(-1, 28*28) X_train = X_train/255.0 X_test = X_test/255.0 X_train[X_train > 0.5] = 1.0 X_train[X_train <= 0.5] = 0.0 X_test[X_test > 0.5] = 1.0 X_test[X_test <= 0.5] = 0.0 y_train = np_utils.to_categorical(y_train, 10) y_test = np_utils.to_categorical(y_test, 10) encoder_m1 = load_model('./trained_model/encoder_m1.h5', custom_objects=custom_objects) decoder_m1 = load_model('./trained_model/decoder_m1.h5', custom_objects=custom_objects) encoder_m2 = load_model('./trained_model/encoder_m2.h5', custom_objects=custom_objects) decoder_m2 = load_model('./trained_model/decoder_m2.h5', custom_objects=custom_objects) X_targets = X_train[9:17] y_targets = y_train[9:17] z1 = encoder_m1.predict(X_targets, batch_size=8) z2 = encoder_m2.predict([z1, y_targets], batch_size=8) fig = plt.figure(figsize=(14, 14)) for i, z in enumerate(z2): ax = fig.add_subplot(8, 11, 11*i+1, xticks=[], yticks=[]) ax.imshow(X_targets[i].reshape(28, 28), 'gray') for j, y in enumerate(np.eye(10)): z1_reconstruct = decoder_m2.predict([y.reshape(1, -1), z2[i].reshape(1, -1)], batch_size=1) x_reconstruct = decoder_m1.predict(z1_reconstruct, batch_size=1) ax = fig.add_subplot(8, 11, 11*i+j+2, xticks=[], yticks=[]) ax.imshow(x_reconstruct.reshape(28, 28), 'gray') plt.savefig('./images/analogy_m1_m2.png')
左1列が元の画像で右10列が固定の元ラベルを変えた画像です.

元画像の筆跡を捉えているのが確認できます.
まとめ
深層学習をわりと自然に生成モデルの枠組みに適用したVAEの再現実験をしました.
最終的に深層学習で画像が生成できると言っても適当な画像が出てきたのでは意味がありません. 如何に狙った画像を従来の人間らしく, もしくは有用な形で出力できるかが大事になります. その点において生成モデルは人間の狙いを組込みやすいモデルなのではないかと思っています.
最後に今回の実装に使ったコードはhttps://github.com/rarilurelo/keras-VAEに置いてあります.
間違っている部分わかりづらい部分ありましたら教えて下さい.